Résumé de section
-
Formation EBAII IFB Niveau 2 : Traitement des Données de Variants, ChIP-Seq et Bulk RNA-Seq du 1 au 6 juin 2025.

Objectifs
La formation s’adresse à des biologistes directement impliqués dans des projets “Next Generation Sequencing” (NGS). Cette édition de l’école aborde les nouveaux enjeux technologiques: elle s’articulera autour de trois ateliers thématiques en session parallèle (bulk RNA-seq, ChIP-seq, variants génomiques/GWAS), et abordera la visualisation et l’intégration des données. L’école vise à approfondir les concepts, à manipuler des outils informatiques avancés et à en interpréter les résultats. Elle est basée sur une alternance de courtes sessions théoriques et d’ateliers pratiques. Les participants bénéficieront d’un tutorat personnalisé pour élaborer leur plan d’analyse, et effectuer les premières étapes de traitement de leurs propres données.
Attention : le tutorat n'a pas pour vocation de réaliser l’analyse complète des données des participants.
Public visé
Cette formation est destinée aux biologistes (ingénieurs, doctorants, chercheurs, enseignants-chercheurs, praticiens…) confrontés à l’analyse de données NGS, et qui ne disposent pas des compétences bioinformatiques suffisantes.
Environnement de travail
L’ensemble de la formation reposera sur l’utilisation du langage R.
Prérequis
Les candidats doivent avoir acquis les compétences enseignées durant l’école de niveau 1, ou équivalent : un niveau de base en ligne de commande, R, et (au choix) bulk RNA-seq, ChIP-seq ou variants génomique/genotypage.
Modalités d’inscription
Date limite de pré-inscription : 1er mars 2025 (sélection des participants : mars 2025). Chaque année la demande dépasse de loin notre capacité d’accueil (40 places), le comité d’organisation sélectionne les participants d’après les informations renseignées dans le formulaire d’inscription. Le degré de maturité du projet scientifique impliquant l’analyse de données de séquençage sera un des critères d’évaluation.
Informations et inscriptions : https://ebaii-ngs-niveau2.pasteur.cloud/
Frais d’inscription : 1000€ HT pour les académiques et EPIC; 2500€ HT pour les industriels.
L’hébergement et la restauration sont inclus. Les frais de transport demeurent à la charge des participants.Coordination scientifique : Erwan Corre (CNRS), Matthias Zytnicki (INRAE), Rachel Legendre (Institut Pasteur), Lucie Khamvongsa-Charbonnier (IFB).
Formateurs et tuteurs :
Matthias Zytnicki Inrae Erwan Corre CNRS Rachel Legendre Institut Pasteur Nadia Bessoltane Inrae Hugo Varet Institut Pasteur Gildas Le Corguillé Sorbonne Université Julien Seiler CNRS Vincent Guillemot Institut Pasteur Lucie Khamvongsa-Charbonnier CNRS Charlotte Berthelier CNRS Pascal Martin INRAE Cathy Philippe CEA Pauline François ANSES Elodie Darbo INSERM Plateformes : IFB core (Ivry-sur-Seine), ABiMS (CNRS/Sorbonne Université, Roscoff).
Coordination administrative : Inserm, IFB, INRAe
Doctorants : Cette école délivre un certificat de présence pour 39H de formation.
Subvention: Une possibilité sera offerte aux agents INRAe et INSERM d’être subventionnés.
Numéro de déclaration de l’Inserm - organisme de formation : 11754553375
Mail contact : ecole-bioinfo@inserm.fr -
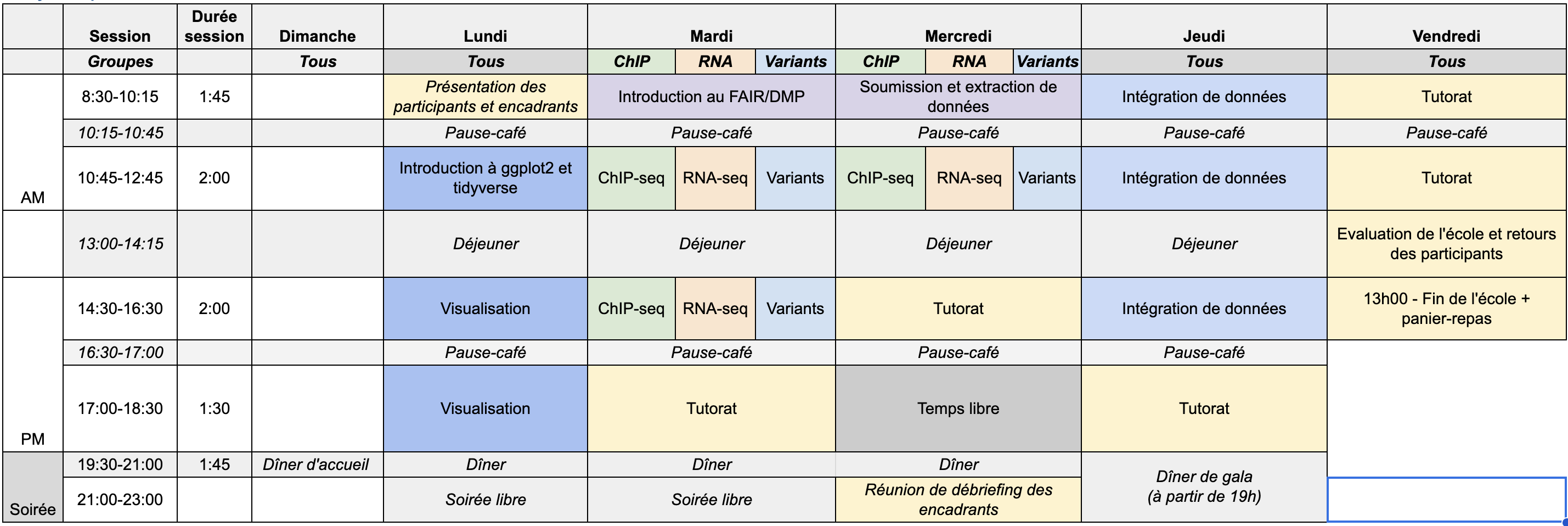
10h15-10h45Horaire Titre Orateur Support Lundi 2 Juin 8h30-9h00 Configuration des ordinateurs pour les participants Erwan Corre
Jean Michel AroumougomSlide ? 9h00-10h15 Présentation de l'école, des participants et institutions
(Aviesan + IFB + Station Roscoff)Tous les participants + intervenants Slide ouverture
Slide participants
Slide intervenants10h15-10h45 Pause café 10h45-12h45 Introduction, compléments et introduction à la manipulation de données avec `dplyr` Vincent Guillemot Slides 14h30-16h30 Introduction à la visualisation avec `ggplot2`, et introduction au R Markdown Vincent Guillemot Slides 16h30-17h00 Pause café 17h00-18h30 Atelier: reproduction d'une figure d'un article scientifique Vincent Guillemot Solution Mardi 3 Juin 8h30-10h15 Introduction au FAIR/DMP Charlotte Berthelier
Pauline FrançoisSlide Mercredi 4 Juin 8h30-10h15 Introduction au FAIR/DMP Charlotte Berthelier
Pauline FrançoisSlide
TP_bash
TP_RJeudi 5 Juin 8h30-10h15 Intégration de données Vincent Guillemot
Cathy PhilippeSlide 10h15-10h45 Pause café 10h45-12h15 Intégration de données Vincent Guillemot
Cathy PhilippeScript 12h45-14h30 Pause déjeuner 14h30-16h00 Intégration de données Vincent Guillemot
Cathy PhilippeSlide
TP16h30-17h00 Pause café 17h00-18hh30 Tutorat Vendredi 6 Juin 09h00-10h15 Tutorat 10h15-10h45 Pause café 11h00-12h00 Tutorat 12h00-12h45 Enquête d'évaluation Matthias Zytnicki, Erwan Corre,
Rachel Legendre,
Lucie Khamvongsa-Charbonnier12h45-13h15 Débriefing et perspectives Tous 13h15 Clôture de l'école -
Horaire Titre Orateur Support Mardi 3 Juin 10h45-12h45 ChIP Pascal Martin,
Elodie DarboSlide
TP12h45-14h30 Pause déjeuner 14h30-16h00 ChIP Pascal Martin,
Elodie DarboSlide
TP16h30-17h00 Pause café 17h00-18h30 Tutorat Mercredi 4 Juin 10h45-12h45 ChIP Slide
TP12h45-14h30 Pause déjeuner 14h30-16h00 Tutorat 16h30-17h00 Pause café 17h00-18h30 Temps libre -
Horaire Titre Orateur Support Mardi 3 Juin 10h45-12h45 RNA Hugo Varet Slide
TP12h45-14h30 Pause déjeuner 14h30-16h30 RNA Hugo Varet Slide
TP16h30-17h00 Pause café 17h00-19h00 Tutorat Mercredi 4 Juin 10h45-12h45 RNA Slide
TP12h45-14h30 Pause déjeuner 14h30-16h00 Tutorat 16h30-17h00 Pause café 17h00-19h00 Temps libre -
Horaire Titre Orateur Support Mardi 3 Juin 10h45-12h45 Rappel Nadia Bessoltane Slide 12h15-12h45 Manipulation de VCF sous R Nadia Bessoltane html
sourcePause déjeuner 14h30-16h30 GWAS Cathy Philippe html
source16h30-17h00 Pause café 17h00-18h30 Tutorat Mercredi 4 Juin 10h45-12h45 GWAS Cathy Philippe Pause déjeuner 14h30-16h30 Tutorat 16h30-17h00 Pause café