Section outline
-


L’Institut Français de Bioinformatique (IFB) organise en partenariat avec iPOP-UP (représenté par EDC) une formation sur les langages de workflows en bioinformatique à destination des bioinformaticien·ne·s et des bioanalystes. La formation abordera les fondamentaux et les fonctionnalités avancées des deux langages Snakemake et Nextflow. Ces outils sont en effet devenus indispensables pour assurer la reproductibilité et l’efficacité des analyses bioinformatiques. La formation sera structurée en trois séquences :
- une journée commune qui abordera les grands principes des gestionnaires de workflow, en particulier dans le domaine de la bioinformatique et en lien avec les infrastructures de calcul de type cluster et cloud proposés au sein de l’IFB
- une journée de session pratique avec 1 atelier snakemake (v.8) et 1 atelier nextflow (DSL2) en parallèle au choix des participants. Nous proposons aux participants qui le souhaitent de travailler sur leur propre workflow dans une approche “Bring your own script” avec l’aide de l’équipe pédagogique.
- une demi-journée "Bring your own script".
Objectifs pédagogiques
A la fin de cette formation les participants :
- auront acquis les bases et connaîtront les fonctions avancées des gestionnaires de workflows
- auront écrit et/ou converti un workflow d’analyse (apporté par le participant ou proposé par l’équipe pédagogique) dans le langage de leur choix (Snakemake v.8, Nextflow DSL2)
- auront exécuté et optimisé leur workflow sur le cluster ou le cloud mis à disposition par l’IFB
Pré-requis
- Maîtrise d’unix et d’un langage de ligne commande (bash ou équivalent)
- Avoir déjà exécuté au moins une analyse avec un gestionnaire de workflow (Snakemake, Nextflow ou équivalent) sans avoir obligatoirement développé le workflow
- Avoir des connaissances sur les environnements de travail type cluster et/ou cloud.
Modalités
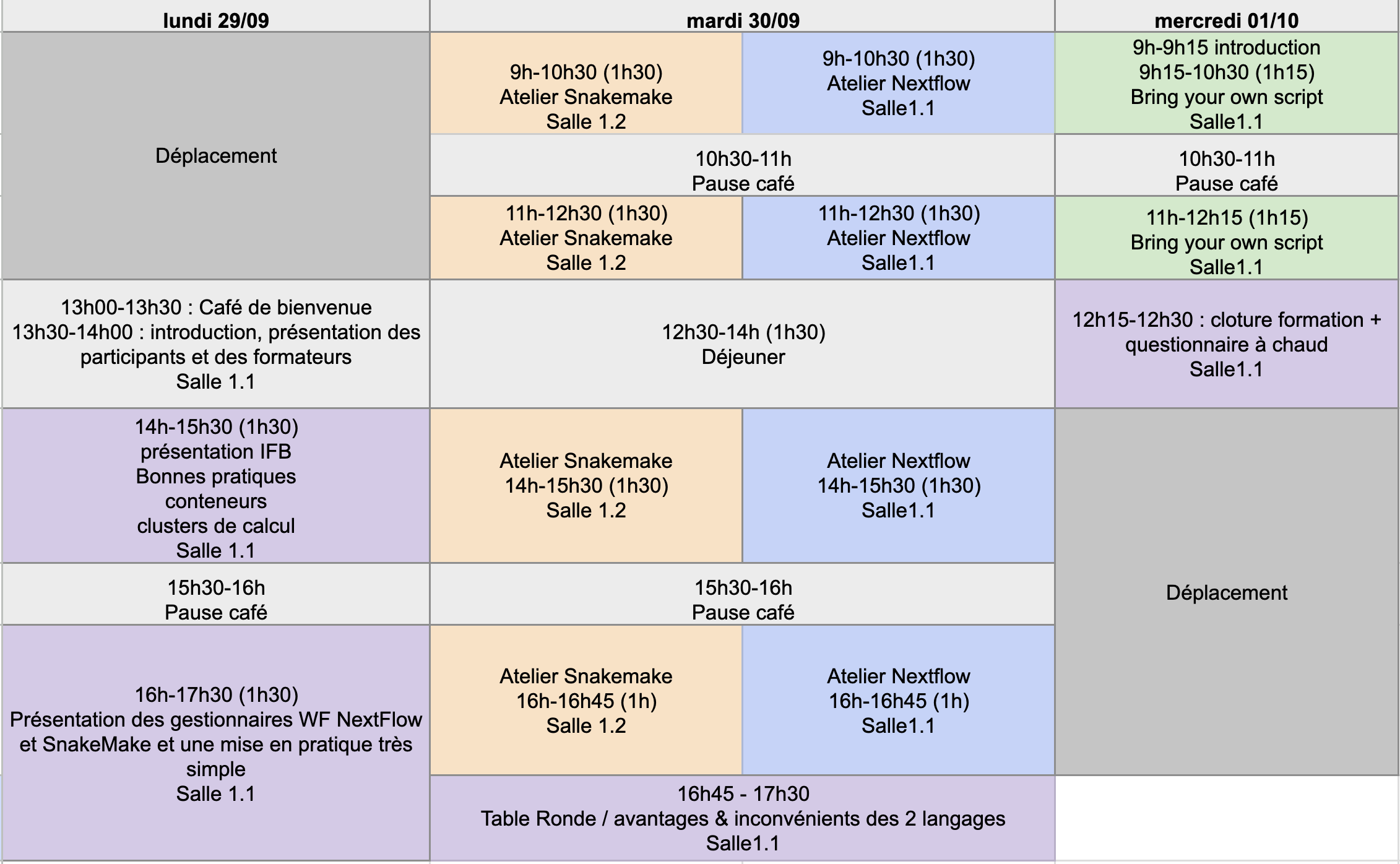
La formation aura lieu du lundi 29 septembre - 13h00 au mercredi 1 octobre 2025 - 12h30 en présentiel à Paris.
Adresse : Institut des Systèmes Complexes Paris Île-de-France, 113 rue Nationale, 75013, Paris, France. Interphone : Sonner à “isc-accueil”.
Métros : Olympiades (M14) and Nationale (M6) Station Vélib : place Nationale.
Les participants devront se munir de leur ordinateur portable.
Tarifs
- Académique : 250 €
- Non-académique : 1000 €
Ces tarifs incluent le repas du midi du jour 2 (plateaux repas) et les pauses café du matin et de l'après-midi
Equipe pédagogique
- Cédric Midoux Migale - INRAE
- Julien Roméjon Institut Curie
- Frédéric Jarlier Institut Curie
- Philippe Hupé Institut Curie
- Quentin Duvert Institut Curie - IFB
- Chloé Quignot BIOI2 - I2BC / CEA / Paris-Saclay
- Pauline Lasserre-Zuber GDEC - UMR INRAE/UCA
- Nadia Goué AuBi - IFB
- Nils Giordano ICAGEN, CRCI2NA
- Julien Seiler IFB
- Manont Connault Migale - IFB
Equipe organisatrice
- IFB: H. Chiapello, L. Jourdren, L. Khamvongsa-Charbonnier
- iPOP-UP représenté par l'unité Epigénétique et Destin Cellulaire: M. Hennion
Date à retenir
Ouvertures des inscriptions : 31 mars 2025
Clôture des inscriptions : 23 mai 2025
Sélection des candidats : juin 2025
Fermeture des inscriptions administratives et mise en paiement : 15 septembre 2025


- une journée commune qui abordera les grands principes des gestionnaires de workflow, en particulier dans le domaine de la bioinformatique et en lien avec les infrastructures de calcul de type cluster et cloud proposés au sein de l’IFB
-
-
Introduction
We will begin with a presentation of the Institut Français de Bioinformatique (IFB) and the resources available to the community. We will then move on to a reminder of best practices for adhering to the FAIR principles and explore how the use of workflow languages and controlled environments aligns with this approach.
We will finish on a short introduction and demo exercise for both NextFlow and Snakemake systems.
-
-
-
-
Atelier Snakemake
In this session, you will follow a guided exercise at your own pace in order to construct a Snakemake pipeline from scratch. This exercise is cut into several parts:
- part A: basic snakemake pipeline
- part B: add extra features
- part C: adapt your pipeline to an HPC environment
- part D: description of a few extra features to bring home
There will also be a quick presentation of a use case with Snakemake. -
-
Atelier Nextflow
In this session, you will learn how to code a Nextflow pipeline from scratch. -
-
-
-
"Bring your own script"
In this session, you will work on your own script in Snakemake or Nextflow.
If you don't have your own script to work on, you can continue the sessions from the previous day if you haven't finished them or try converting the following bash script into a Snakemake or Nextflow pipeline.
Input data can be found here: -
-