Résumé de section
-
Formation EBAII IFB Aviesan Niveau 1 : Traitement des Données de Variants, ChIP-Seq, Bulk RNA-Seq, et Single-Cell RNA-Seq
Description : La formation EBAII IFB Aviesan de niveau 1 propose une expérience d'apprentissage intensive conçue pour les biologistes, qu'ils soient ingénieurs, doctorants, chercheurs, enseignants-chercheurs ou praticiens, qui sont confrontés à l'analyse de données NGS (Next-Generation Sequencing) mais qui ne disposent pas encore des compétences bioinformatiques nécessaires, ou qui cherchent à renforcer leurs compétences existantes.
Contenu : Cette formation est structurée autour d'une combinaison de sessions théoriques et d'ateliers pratiques. Les participants auront l'occasion d'explorer diverses thématiques, notamment le traitement de données de variants, ChIP-Seq, Bulk RNA-Seq, et Single-Cell RNA-Seq. De plus, ils recevront une introduction aux technologies "long reads".
Objectifs généraux:
- Acquérir une compréhension approfondie des concepts liés à l'analyse de données NGS.
- Maîtriser les outils informatiques nécessaires pour effectuer ces analyses.
- Interpréter les résultats des analyses de données NGS.
Durée : La formation s'étend sur une période de cinq jours, du dimanche à 18h au vendredi à 14h, offrant aux participants une immersion complète dans le domaine de l'analyse de données NGS.
Environnement de Travail : Les participants seront exposés à un environnement de travail basé sur l'utilisation de commandes en ligne via un terminal Linux et du langage de programmation R. Aucune connaissance préalable de ces environnements n'est nécessaire, mais les participants seront encouragés à suivre une autoformation en ligne avant le début de la formation pour faciliter leur prise en main de ces langages. La formation approfondira progressivement l’usage de ces environnements au fil des sessions thématiques.
Tutorat Personnalisé : Les participants bénéficieront d'un tutorat personnalisé pour élaborer leur plan d'analyse et pour effectuer les premières étapes du traitement de leurs propres données ou de celles de leur équipe. Il est important de noter que le tutorat ne vise pas à réaliser l'analyse complète des données des participants, mais plutôt à les guider dans le processus.
Langue : Tous les cours de cette formation seront dispensés en français.
Lieu : Les cours se dérouleront à Roscoff, à l'Hôtel de France.
Frais d'Inscription : Les frais d'inscription s'élèvent à 1000€ HT pour les participants académiques et EPIC, et à 2500€ HT pour les participants industriels. Ces frais comprennent l'hébergement et la restauration, mais les frais de transport restent à la charge des participants.
La formation EBAII IFB Aviesan Niveau 1 offre une opportunité unique pour les professionnels de la biologie de développer leurs compétences en bioinformatique et d'acquérir une compréhension approfondie de l'analyse de données NGS, le tout dans un environnement d'apprentissage intensif et pratique.
-
-
Introduction à Linux
-
Introduction à

-
Présentation Cluster
-
Croisement de données
-
Programme de l'atelier ChIP-seq
Design expérimental, Contrôle qualité Stéphanie Le Gras Alignement Stéphanie Le Gras Contrôle qualité des alignements, visualisation Tao Ye Peak-calling Tao Ye Analyse des motifs Morgane Thomas-Chollier Annotation des pics Elodie Darbo Workflow et conclusion Denis Puthier -
-
Statistical aspects for Differential Expression Analysis and experimental design in RNASeq
-
Analyse différentielle : exercices pratiques avec SARTools
- Script RNASeq_DEA.r avec le paramétrage et étapes pour l'analyse différentielle
- Fichier target.txt résumant le design experimental
- dossier raw avec des tableau de comptes obtenus en sortie du pipeline bioinfo
-
Analyses d'enrichissement
- Script TP_GSEA_TP.R avec le paramétrage et étapes pour faire :
- homogénéisation de l'annotation des gènes pour être compatible avec les bases de données de signatures (gene sets)
- Over Representation Analysis (ORA)
- Gene Set Enrichment Analysis (GSEA)
- Fichier KOvsWT.complete.txt, issu du TP "Expression Différentielle"
- Script TP_GSEA_TP.R avec le paramétrage et étapes pour faire :
-
Assemblage de Novo
-
-
Programme de l'atelier scRNA-seq
Analyses primaires Technologies, qualité et alignement
Visualisation IGV
Construction de la matrice de comptageAnalyses secondaires Préparation de la matrice de comptage
Normalisation et visualisation
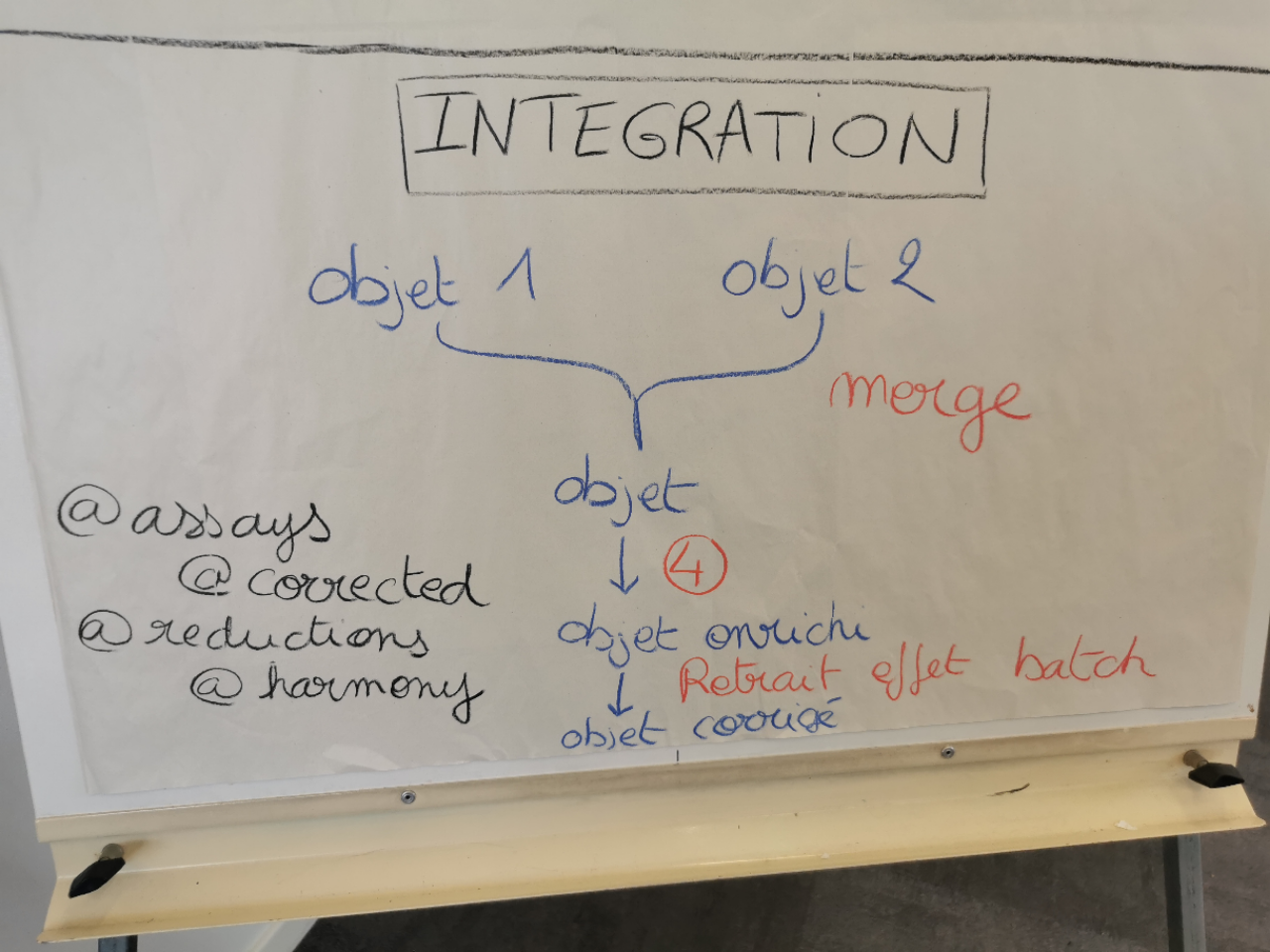
Analyses des gènes et annotation des types cellulairesPour aller plus loin Intégration de données
Inférence de trajectoireConclusion
Ressources techniques (JupyterHub, RStudio, R, Rmarkdown)
Description d'un objet Seurat
Équipe :
Thibault Dayris
Émilie Drouineau
Bastien Job
Nathalie Lehmann
Sophie Lemoine
Eulalie Liorzou
Rémi Montagne
Audrey Onfroy
Morgane Thomas-Chollier-
-
- Normalisation et scaling de la matrice de comptage
- Réduction de dimensions
- Visualisation

-
- Analyses d'expression différentielle
- Analyses d'enrichissement en signatures
- Annotation des types cellulaires

-
Ce document présente brièvement des notions sommaires de l' "après", du point de vue :
- des analyses
- des technologies / applications techniques
3.7 Mo · Déposé le 9 nov. 23, 12:11 -
Autres documents
-


Ce PDF détaille les éléments principaux de la structure d'un objet Seurat (v4)
- sous forme textuelle
- sous forme graphique
669.3 Ko · Déposé le 8 nov. 23, 23:59 -
1.2 Mo · Déposé le 7 nov. 23, 09:10
-
2.7 Mo · Déposé le 7 nov. 23, 11:38
-
1.6 Mo · Modifié 7 nov. 23, 09:03
-
6.8 Mo · Déposé le 6 nov. 23, 21:58
-
Modifié 7 nov. 23, 09:06
-
1.9 Ko
-